Archivos Mensuales: julio 2011
Mapas de proteínas
Comenzamos con este artículo ya a entrar en materia y dejamos atrás los artículos básicos sobre aminoácidos, enlaces peptídicos y estructura de proteínas.
En el análisis global de los componentes de un proteoma nos podemos encontrar a grandes rasgos con dos estrategias:
a) Convencional: separar todas las proteínas de la muestra (por electroforesis en dos dimensiones en general) e identificar aquellas de interés por espectrometría de masas.
b) A gran escala (“shot-gun”): la muestra es tratada con una enzima proteolítica y los péptidos resultantes se separan por HPLC acoplada a un sistema de espectrometría de masas. Esto permite la identificación de todas las proteínas presentes en la mezcla.
En este post vamos a ver la primera de ellas, la estrategia convencional, la cual se basa en el empleo de la electroforesis bidimensional.
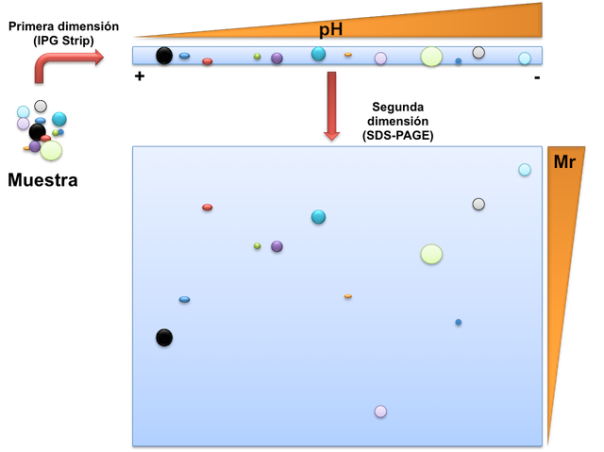
La electroforesis bidimensional es una técnica de alta resolución para separar mezclas de proteínas muy complejas. La base de su gran poder de resolución es la bidimensionalidad, es decir, que las proteínas son separadas ortogonalmente al aplicar dos métodos físicos distintos. Una primera dimensión separa las proteínas en un gel de acrilamida con gradiente de pH por su punto isoeléctrico (isoelectroenfoque, del inglés isoelectric focusing IEF). En la segunda dimensión las proteínas son separadas por su masa molecular por electroforesis en poliacrilamida en presencia de SDS (SDS-PAGE). Tras teñir el gel, las proteínas aparecen como manchas circulares. (www.bioinformatica.uab.es).
El punto isoeléctrico (pI) de una proteína es el pH al que su carga neta es cero, por lo que no se desplaza en un campo eléctrico. Al introducir una mezcla de proteínas en un gel con un gradiente de pH, sus extremos N y C terminal (recordadlo en este post) y sus residuos ionizables captan o liberan protones de acuerdo con el pH, por lo que su carga eléctrica global dependerá del pH del entorno. Al aplicar un campo eléctrico, todas las moléculas con carga neta positiva serán atraídas hacia el cátodo, y aquellas con carga neta negativa hacia el ánodo. A medida que las moléculas de proteína se acercan a su pI van perdiendo carga eléctrica, hasta llegar al valor de pH en que su carga neta sea cero y dejen de moverse. Se dice entonces que las proteínas han enfocado, pues todas las moléculas de una proteína inicialmente dispersas por todo el gradiente de pH se han concentrado en un solo punto, que corresponde a su pI; de aquí el nombre de IEF. El enfoque se realiza a voltajes muy altos, de hasta 10000V, y en condiciones desnaturalizantes (ej. 8M urea) para obtener la máxima resolución.
En sus inicios, a finales de los 70, estos gradientes de pH se generaban con una mezcla compleja de pequeños polímeros con gran capacidad tampón cerca de su pI, o carrier ampholytes (CA), aunque tenían problemas como distorsión del gradiente de pH, variabilidad entre geles y gran complejidad química. Hoy, los anfolitos han sido sustituidos por las tiras IPG (gradientes de pH inmovilizados en un soporte plástico) generados por la copolimerización, junto a acrilamida y bisacrilamida, de un gradiente de monómeros de acrilamida derivatizados con grupos ácidos o básicos cuya unión covalente a la matriz impide la distorsión del gradiente. Las tiras IPG tienen muchas ventajas: i) tolerancia a presencia de sales en la muestra, ii) mayor capacidad de carga, iii) numerosos gradientes de pH (incluso muy estrechos, de <1 unidad), iv) gran disponibilidad comercial (GEHealthcare, Bio-Rad Laboratories).
La segunda dimensión consiste en una separación SDS-PAGE convencional. La tira IPG se deposita sobre el gel SDS-PAGE y se lleva a cabo la electroforesis.
Tras realizar la segunda dimensión hay que teñir los geles. Hay muchos métodos de tinción de proteínas en geles de poliacrilamida pero sólo algunos se usan en 2-DE. El más usado durante muchos años ha sido la tinción con plata, por su alta sensibilidad, pues revela menos de 1 ng de proteína. Sin embargo, la plata no es una tinción a punto final y su rango lineal es muy estrecho. Por ello, ha sido sustituido por métodos de tinción con colorantes fluorescentes como el Sypro Ruby que, aunque mucho más caros, tienen una sensibilidad comparable a la de la plata y un rango dinámico lineal de hasta tres órdenes de magnitud, óptimos para estudios cuantitativos.
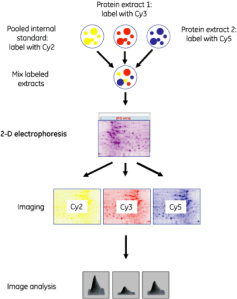
Como desarrollo de la 2-DE ha surgido una nueva tecnología que saca todo el provecho de la 2-DE y la tinción con fluoróforos: la técnica DIGE (Difference Gel Electrophoresis). Esta técnica corrige los problemas de reproducibilidad, detección de manchas y posterior análisis de la 2-DE convencional. Se basa en las propiedades de 3 tipos de fluoróforos de la familia de las cianinas (CyDye: Cy3, Cy2, Cy5), que emiten luz a distinta longitud de onda. Cada muestra se marca con uno de ellos y, tras el marcaje, se mezclan antes del isoelectroenfoque; se separan hasta 3 muestras diferentes (2 problema y 1 control generado al mezclar ambas, ó control interno) en un sólo gel, eliminándose la variación entre geles y asegurando que los efectos de sobre/sub-expresión visualizados en el gel se deben sólo a cambios biológicos. Así, cada mancha de proteína puede ser comparada fácilmente en distintas condiciones de manera semicuantitativa.
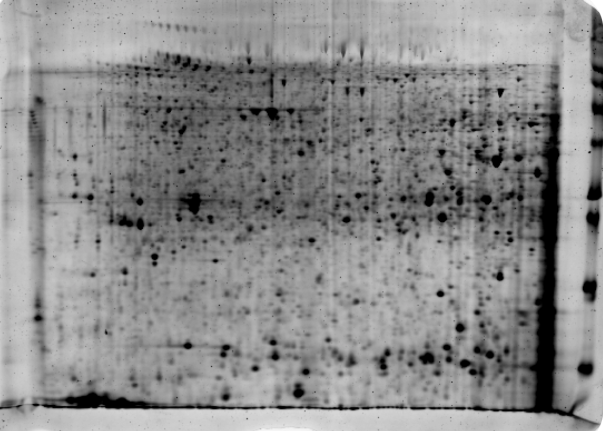
Al final de todo el proceso de la electroforesis bidimensional vamos a obtener unos mapas de las proteínas que se estaban expresando en un determinado momento en el organismo/tejido/cultivo tan chulos como este que os muestro a continuación y que obtuvo el que os escribe durante el desarrollo de su Tesis Doctoral:
Y aquí es donde comienza el trabajo de espectrometría de masas y bioinformática, pero eso ya es otra historia que os contaré otro día…